Koronawirusy patogenne dla trzody chlewnej – wirus zakaźnego zapalenia żołądka i jelit
Wirusowe zapalenie żołądka i jelit (ang. transmissible gastroenteritis – TGE) zostało opisane po raz pierwszy w 1946 roku w Stanach Zjednoczonych (1). Od tego czasu wirus wywołujący TGE wykrywano praktycznie na całym świecie. Chorobę wywołuje wirus zakaźnego zapalenia żołądka i jelit (ang. transmissible gastroenteritis virus – TGEV) należący do rodziny Coronaviridae, rodzaju Alphacoronavirus. Poza tym w obrębie rodzaju Alphacoronavirus zidentyfikowano jeszcze trzy koronawirusy odpowiadające za infekcje związane z układem pokarmowym u świń: koronawirusa płucnego świń (PRCV), wirusa epidemicznej biegunki świń (PEDV) oraz koronawirusa zespołu ostrej biegunki świń (SADS-CoV) (2). W przebiegu TGE obserwuje się intensywną biegunkę, wymioty i wysoką śmiertelność wśród prosiąt w wieku poniżej dwóch tygodni (3). Zakażenie wspomnianymi wirusami wywołuje u trzody chlewnej chorobę o identycznym obrazie klinicznym, dlatego do wykrywania TGEV ważna jest diagnostyka różnicowa oparta na badaniach laboratoryjnych.
Czynnik etiologiczny TGE
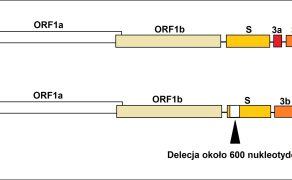
Materiał genetyczny TGEV ma postać RNA o dodatniej polaryzacji, a długość genomu wynosi około 28,5 tysięcy par zasad (4, 5). Genom zawiera tzw. regiony 3` i 5` nieulegające translacji oraz dziewięć ramek odczytu (ang. open reading frame – ORF) pełniących różne funkcje (2). Dwie trzecie genomu TGEV zawiera dwa duże ORF-y, 1a i 1b, kodujące dwie niestrukturalne proteiny, które kierują replikacją genomu i transkrypcją. Pozostała część genomu zawiera ORF-y określające białka strukturalne i niestrukturalne: białko kolca (S), ORF3, białko otoczki (E), glikoproteinę transmembranową (M) i nukleoproteinę (N).
Opisana struktura genomu jest typowa dla wszystkich koronawirusów (2). Białko kolca w dużym stopniu odpowiada za zjadliwość wirusa i odgrywa znaczącą rolę w procesie wnikania wirusa do komórek gospodarza (6). Mutacje w obrębie tego białka mogą wpływać na zmiany w zakresie zjadliwości i tropizmu TGEV (6, 7). Na podstawie sekwencji nukleotydowej ORF3 wśród szczepów TGEV można wyróżnić klaster Miller i klaster Purdue. ORF3 TGEV zawiera sekwencje kodujące niestrukturalne proteiny 3a i 3b. Delecje w obrębie sekwencji kodującej proteinę 3a mogą powodować zmiany w zjadliwości i tropizmie wirusa, a także skutkować całkowitą utratą ORF3a z genomu (8).
Genom TGEV zawiera także ORF7 (6) (ryc. 1).
TGEV jest wrażliwy na wysoką temperaturę, światło słoneczne i promieniowanie ultrafioletowe (7, 9, 10). Pozostaje infekcyjny w płynnych odchodach ponad 8 tygodni w temperaturze 5°C, a w temperaturze 35°C – tylko przez 24 godziny (11). Jego stabilność w niskich temperaturach jest związana z częstszymi wybuchami TGE w okresie zimowym. Wirus jest skutecznie inaktywowany 0,03-proc. formaliną, 1-proc. roztworem fenolu i aldehydu, nadtlenkiem wodoru, środkami dezynfekcyjnymi na bazie jodu, chloroformem czy czwartorzędowymi solami amoniowymi (7).
Występowanie TGEV na świecie
Choroba o przebiegu i cechach charakterystycznych dla TGE została opisana po raz pierwszy w 1935 r., chociaż sam czynnik etiologiczny został zidentyfikowany ponad 10 lat później (1, 12). TGEV prawdopodobnie krążył wcześniej w populacji trzody chlewnej bez znaczącego wpływu na produkcję i zdrowie, stając się ważnym czynnikiem patogennym świń wraz z intensyfikacją produkcji (12). Wirus ten był izolowany na całym świecie w ciągu kolejnych 20 lat od pierwszej identyfikacji. TGE znajduje się także na liście chorób, których wystąpienie należy zgłosić do Światowej Organizacji Zdrowia Zwierząt (WOAH – World Animal Health Organisation).
Według danych z systemu WAHIS (World Animal Health Information System) choroba wystąpiła trzy razy wśród państw członkowskich: w Polinezji Francuskiej i Peru w 2011 r. oraz w Argentynie w 2014 r. (13). Badania dotyczące występowania TGEV są przeprowadzane w Stanach Zjednoczonych i Chinach. W Stanach Zjednoczonych ogólna prewalencja wynosiła 2,3% (667/29 397 próbek kału i tkanek jelit), wahała się od 0,1% do 6,8% w latach 2008-2016, osiągając najniższą wartość w latach 2015-2016 (14). W Chinach materiał genetyczny wirusa wykryto w 0,7% (21/2987) próbek kału pobieranych w latach 2012-2018 (15). Odnotowuje się także występowanie zjadliwych szczepów TGEV w tym kraju. W 2020 r. wyizolowano szczep JS2012 (6).
Zakażenie eksperymentalne tym szczepem skutkowało 100-proc. śmiertelnością nowo narodzonych prosiąt (6). Kolejny zjadliwy szczep TGEV, SC2021, wyizolowano w 2023 r. (16). Sekwencja genomowa wirusa wykazała delecję 81 nukleotydów w ORF3a, co skutkowało brakiem ekspresji niestrukturalnej proteiny 3a oraz prawdopodobnie zwiększeniem zjadliwości i zmianą w powinowactwie wirusa do komórek układu oddechowego. W trakcie badania nie zauważono znaczących objawów z układu oddechowego, jednak szczep SC2021, oprócz zmian charakterystycznych dla TGE, powodował także poważne uszkodzenie tkanki płucnej prosiąt. TGE u prosiąt zakażonych tym szczepem przebiegało z silnie wyrażonymi objawami i wysoką śmiertelnością (16).
Brak jest najnowszych badań epidemiologicznych w aspekcie zakażeń TGEV w wielu krajach, ale mimo to wydaje się, że jest on prawie nieobecny w Europie (17). Zgodnie z obowiązującą Ustawą z dnia 11 marca 2004 r. o ochronie zdrowia zwierząt oraz zwalczaniu chorób zakaźnych zwierząt TGE podlega w Polsce obowiązkowi rejestracji. Aktualnie brak oficjalnych zgłoszeń dotyczących występowania TGEV w Polsce.
W ostatnich latach odnotowuje się koinfekcje TGEV z innymi, m.in. PEDV oraz PRCV (18-20). Wymienione wirusy należą do alfa-koronawirusów patogennych dla świń, a koinfekcje wśród tej grupy są częste. Notowane są rekombinacje pomiędzy TGEV a PEDV, co może skutkować powstawaniem szczepów TGEV o nowych właściwościach, w tym wirusów o zwiększonej zjadliwości (21).
Galeria
POSTĘPOWANIA
w weterynarii