Diagnostyka laboratoryjna chorób świń. Cz. II. Pułapki w interpretacji wyników badań laboratoryjnych
Powyższa kwestia ma bardzo duże znaczenie dla interpretacji wyników tego badania. Pozytywne wyniki izolacji pokazują obecność dostatecznej ilości żywych bakterii danego gatunku w materiale badawczym. Natomiast wynik negatywny (brak izolacji) nie jest równoznaczny z tym, że bakterii chorobotwórczych w danej próbce nie było. Przyczynami tego mogły być: nie do końca dobrze ukierunkowana metoda, śmierć bakterii w materiale podczas transportu lub to, że było ich na tyle mało, że izolacja nie była skuteczna. Dlatego też wykluczenie danego gatunku bakterii jako czynnika sprawczego na podstawie negatywnego wyniku badania bakteriologicznego powinno być formułowane bardzo ostrożnie.
Kolejną ważną sprawą dotyczącą badania bakteriologicznego jest identyfikacja, czy dany szczep jest chorobotwórczy. W przypadku wielu świńskich patogenów istnieje duże zróżnicowanie, jeśli chodzi o potencjał do wywołania choroby u świń.
To, czy dany izolat jest silnie chorobotwórczy, zależy np. od serotypu, jak w przypadku Actinobacillus pleuropneumoniae czy Streptococcus suis lub posiadanych czynników zjadliwości (wirulencji), takich jak: otoczki, endo- i egzotoksyny, fimbrie i rzęski, białka błony zewnętrznej, lipopolisacharyd. Na przykład wynik badania, w którym stwierdzono izolację bakterii Escherichia coli z wymazów z odbytu od prosiąt z biegunką, jest bezwartościowy, bowiem bakterie te są naturalnym składnikiem flory bakteryjnej.
Dopiero dalsze informacje na temat charakterystyki wyizolowanych szczepów pozwalają na precyzyjne określenie ich roli w procesie chorobowym. Stwierdzenie: „W posiewach z wymazów z odbytu stwierdzono umiarkowany wzrost beta-hemolitycznych szczepów Escherichia coli” również nie jest wyczerpujące. Nie zawsze patogenne szczepy E. coli będą wykazywały hemolizę. Część szczepów (około 30%) będących czynnikami sprawczymi biegunki u prosiąt będzie rosła na agarze z krwią bez hemolizy. Dlatego ważne jest określenie posiadania przez dany szczep specyficznych fimbrii adhezyjnych (F4, F5, F6, F41) i zdolności do wytwarzania enterotoksyn (LT, Sta, Stb). Również w przypadku innych gatunków bakterii często konieczna jest identyfikacja czynników zjadliwości.
Metody biologii molekularnej
Obecnie bardzo często w diagnostyce laboratoryjnej chorób świń wykorzystywane są metody biologii molekularnej. Najczęściej jest to reakcja łańcuchowa polimerazy (PCR – ang. polymerase chain reaction) oraz jej modyfikacja, czyli PCR w czasie rzeczywistym (real-time PCR). Metoda ta charakteryzuje się wysoką czułością umożliwiającą wykrycie nawet pojedynczych cząsteczek kwasu nukleinowego danego patogenu.
Każdy test PCR ma określoną granicę wykrywalności, czułość i specyficzność analityczną.
W przypadku interpretacji wyników konwencjonalnego PCR, w którym stwierdzana jest obecność materiału genetycznego danej bakterii czy wirusa, należy pamiętać o tym, że jest to badanie jakościowe, które z dużą czułością może potwierdzić bądź wykluczyć obecność w materiale danego patogenu.
Niestety, nie mówi nam ono nic o tym, jak dużo danego patogenu znajduje się w badanym materiale. Ma to znaczenie, zwłaszcza kiedy jako czynnik sprawczy choroby podejrzane są bakterie warunkowo chorobotwórcze. Na przykład stwierdzenie obecności w próbce kału prosiąt materiału genetycznego Clostridium perfringens typu A jest mało przydatne w rozpoznaniu choroby, bowiem bakterie z tego gatunku są naturalnym składnikiem flory bakteryjnej przewodu pokarmowego świń. Dopiero stwierdzenie, że ilość tych bakterii w kale jest dużo większa niż u zdrowych świń, może sugerować, że są one przyczyną problemu. Jednak żeby to stwierdzić, potrzebny jest wynik badania real-time PCR, qPCR (ilościowy PCR – quantitative PCR), które ze względu na specyfikę działania pozwala na oszacowanie ilości produktu na początku reakcji, co jest niemożliwe w konwencjonalnej metodzie PCR.
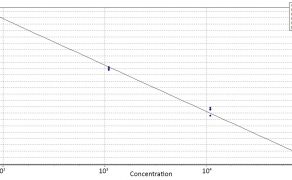
Upraszczając, qPCR pozwala na stwierdzenie ilości patogenu znajdującego się w próbce. Jednak laboratoria, mimo tego, że badają próbki metodą qPCR, rzadko umieszczają w wyniku konkretne wartości, np. liczbę bakterii w ml próbki. Zamiast tego najczęściej dostajemy tzw. wartość Ct (ang. threshold cycle), która oznacza kolejny numer cyklu PCR, w którym aparat wykrył emisję określonej fluorescencji z probówki reakcyjnej. Im ten numer jest niższy, tym więcej wykrywanego fragmentu DNA/RNA znajdowało się w próbce.
Zazwyczaj wartość Ct próbki, którą uznaje się za dodatnią, waha się w granicach 35-39, co zależy już od konkretnej metody i patogenu. Próbki o takiej wartości Ct zawierają bardzo mało materiału genetycznego danego wirusa czy bakterii, czasami są to nawet pojedyncze kopie genów. Należy jednak pamiętać, że wartości Ct nie są wyrażane na skali liniowej tylko na skali logarytmicznej (podobnie jak w przypadku mierzenia pH czy natężenia dźwięku w decybelach). Zakładając, że Ct 36 oznacza zawartość w danej próbce 10 komórek bakteryjnych, Ct równe 25,2 będzie już oznaczało wykrycie ich 10 000, natomiast przy Ct o wartości 18,2 tych komórek będzie już 1 milion w badanym materiale.
Jak widać, różnice liczbowe dotyczące Ct nie oddają tego, jak dużo patogennych drobnoustrojów znajduje się w próbce. Na powyższym przykładzie widać, że zmiana Ct z 36 na 25,2 (różnica liczbowa – 10,8) oznacza wzrost ilości bakterii w próbce (10 vs 10 tys.), ale wciąż są to stosunkowo niskie poziomy. Z kolei różnica liczbowa – 7 między Ct 25,2 a 18,2 wygląda na mniejszą, ale rzeczywista różnica w ilości bakterii w próbce jest ogromna (10 tys. vs 1 mln). Warto o tym pamiętać, kiedy porównujemy wartości Ct otrzymane z laboratorium.
Galeria
Mogą zainteresować Cię również
POSTĘPOWANIA
w weterynarii